
基因编辑技术近年来取得了长足的发展,其中CRISPR技术作为一种革命性工具,已被广泛应用于各类基因操作。然而,CRISPR系统中的核心蛋白,如Cas9和Cas12,尽管具有效率高、适用范围广等优势,但其较大的分子体积限制了在某些应用中的灵活性和有效性。针对这一问题,科学家们一直在探索更为紧凑、高效的基因编辑工具。近日,《Nature Methods》发表了一项来自苏黎世大学的关于TnpB系统的最新研究,该系统不仅体积小巧,而且具备强大的基因编辑能力,为基因编辑技术带来了新突破。

TnpB是一类由转座子编码的蛋白,被认为是CRISPR系统的前身。与CRISPR效应蛋白Cas9和Cas12相比,TnpB具有体积更小的优势。最初,Deinococcus radiodurans (ISDra2) TnpB系统被发现可用于哺乳动物细胞中的基因编辑,它通过一种称为ωRNA的RNA分子引导,结合靶标DNA并进行切割,类似于CRISPR系统中的guide RNA(gRNA)功能。
首先,研究团队通过对TnpB的密码子和核定位信号(NLS)进行优化,开发出了一个新版本的TnpB,称为TnpBmax。这一优化版本大幅提升了在哺乳动物细胞中的基因编辑效率,平均提高了4.4倍。

文章指出,在TnpB系统中,有两大限制其效率的因素,针对这两个方向,研究人员分别给出不同的解决方法。
No.1
《ωRNA的设计》
ωRNA的设计是决定编辑效率的关键。研究团队通过建立一个包含11,188个靶标位点的实验库,详细分析了ωRNA的设计原则,发现TnpBmax偏好靶向嘌呤(A和G)富集的序列,尤其是ωRNA的前12个碱基,不能容许任何错配。这种设计会使ωRNA具有高度的靶向特异性,有助于减少脱靶效应,提高基因编辑的安全性。

No.2
《TnpB系统对TAM的依赖性》
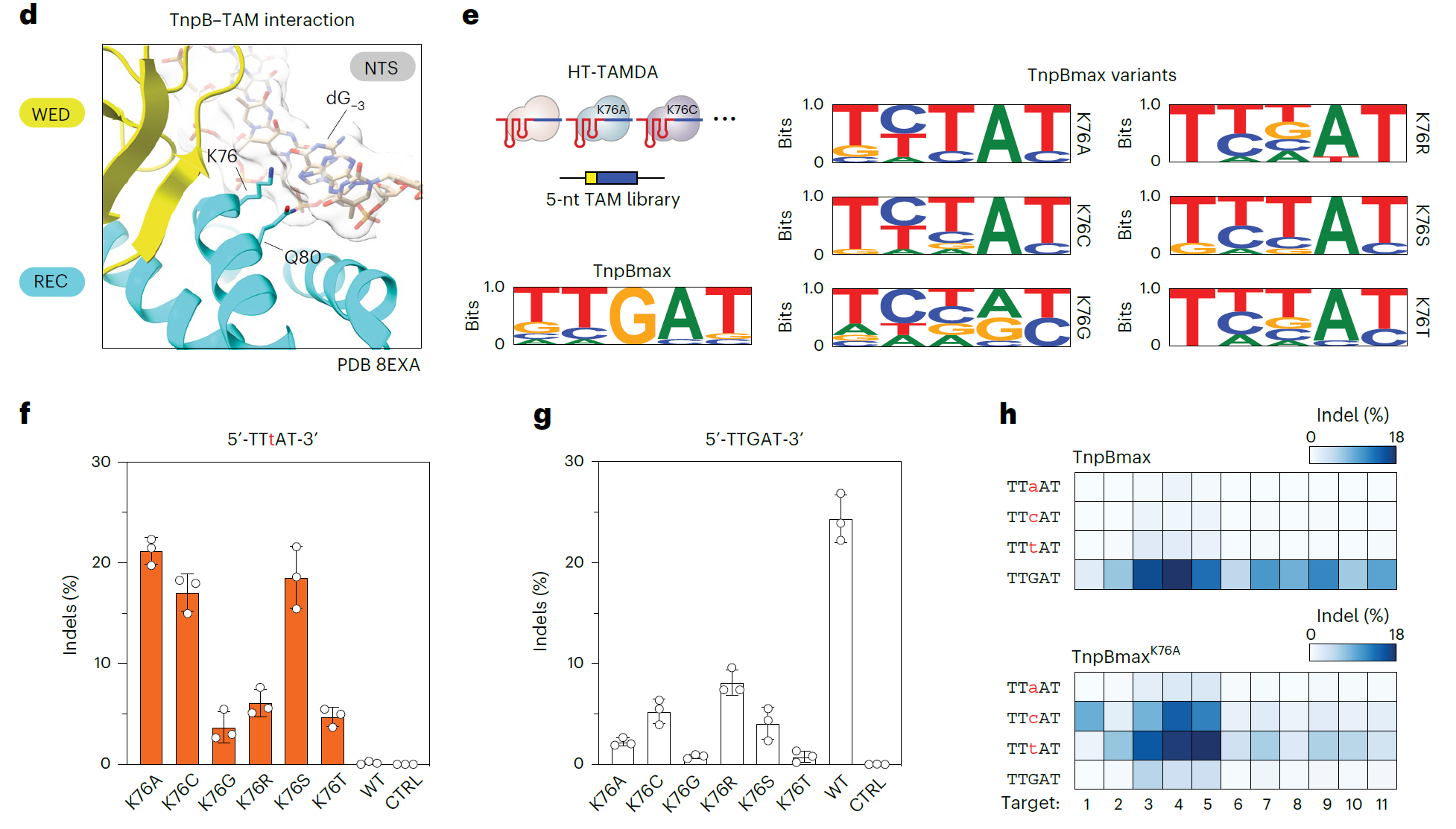
对TAM(靶标相邻基序)的依赖会限制系统的靶向范围,为了克服这一限制,研究团队在TnpB的K76位点引入了突变,并生成了多个能够识别非典型TAM序列的TnpB变体。其中,TnpBmaxK76A表现出对TAM序列5’-TTtAT的高度识别能力。通过这一突变,研究人员成功地将TnpB的靶标范围从原本的5’-TTGAT拓展到了5’-TYKAT,该基序在基因组中出现的频率比TTGAT高出四倍,大大增强了TnpB的应用灵活性。
小巧的体积使得TnpB和ωRNA可以被同时装载到AAV(腺相关病毒)载体中,实现体内基因编辑。研究人员通过在小鼠体内实验发现,使用AAV递送TnpBmax可以实现高达75.3%的基因编辑效率,特别是在小鼠的肝脏和大脑中表现出优异的编辑效果。这一结果表明,TnpB系统不仅在体外实验中表现出色,还具备极大的体内应用潜力。

TnpB系统作为一款超紧凑的基因编辑工具,具备高效、特异性强、靶向范围广等优点,特别是在体内基因编辑中的应用前景广阔。特别值得一提的是,为了简化ωRNA的设计难度,研究团队开发了一款深度学习模型TEEP,为TnpB的设计和应用提供了更为精准的工具,显著减少实验成本和时间。
这篇论文的研究标志着基因编辑技术的又一次重要突破,为未来在基因治疗、疾病研究等领域的应用奠定了坚实基础。欢迎通过下方链接阅读原文。
原文阅读:https://doi.org/10.1038/s41592-024-02418-z

